名古屋でおすすめの博物館
弘前市の果樹園と水田の分布図を作成する
業務で、弘前市のりんご畑と水田の分布図を作成することとなりました。
電子情報の整備された今なら簡単にりんご畑の図くらい書けるだろうと甘くみていました。リンゴ畑のデータがなかったのです。ならば、果樹園ならすぐに手に入るだろうとおもいました。こちらも市内での分布を示すような細かいメッシュデータがありません。そこで、ふと思いつきました。「2万5000分の1地図に出てくるような果樹園マークをポイントにしていけばいいのでは?」ということです。弘前市の果樹園はりんごが主体であることは間違いありません。ほかにも含まれるものの、今考えられる方法はそれしかありませんでした。
次は作業手順です。まず、QGIS上に基盤地図情報から弘前市を表示させました(これは国土地理院のウェブサイトやQGISの解説本を参照ください)に説明があります。そして、ひたすら果樹園マークをポイントデータにしてゆきました。

ポイントデータはシェイプファイルの1種です。QGISメニューのレイヤー>レイヤーの作成>新しいシェイプファイルレイヤーと選び、点(ポイント)が選ばれていることを確認します。あとは、点を打ってはidを順番に入れてゆくだけです。100,200,300・・・と、このあたりで自身の計画の無謀さに気付き始めます。「けっこう多いな・・・」。「1000、2000・・・」これは危険な作業だと気付いたときにはすでに時遅しです。途中で保存し忘れて400ポイント失う事件などもあり、その後は100ポイントずつで保存するようにしました。のべ1週間の作業となり、最終的12,000ポイントとなりました。弘前には一面のリンゴ畑が広がっているのです!
そして、水田は既存データから入手できます。国土交通省のウェブ(下記)で提供されている土地利用細分メッシュデータです。
しかし、このデータはxml形式でそのままだとQGISでは読めません。そこでxmlからshpへの変換ツール(ksjツール)も提供されていたのであわせてダウンロードしてそこから変換を試みました。・・・しかし、うまくいきませんでした。QGISのプラグインで”fgddemImporter”というものが”Minoru Akagi”さんによって提供されています。
こちらを利用したところうまく読み込めました。
しかし、土地利用細分メッシュデータは青森県で4つくらいと大きなデータです。そこで、弘前市の範囲を指定する必要があります。まず、先ほどと同じ国土数値情報のサイトから、行政区域を選択して青森のデータをダウンロードします。そして、やはりfgdemImporterで・・・と思ったら、今度はうまくいきません。こちらはksjツールで変換できました。なぜだか私にはわかりません。
土地利用細分メッシュデータも弘前市以外の部分をまず削除します。レイヤを選択してから、ベクタの編集モード(えんぴつマークをクリック)にして、範囲を選択します。そして、選択物の削除を選ぶと削除できます。削除する量が多いとフリーズしたように見えますし、時としてほんとうにフリーズします。適度な量ずる削除しましょう。


市町村データのほうも同様です。読み込んだ青森県各市町村のデータのうち弘前市だけを抜き出します。シェイプファイルなので、属性テーブルを開いて弘前市以外のセルを削除してゆけばよいです。
そして、弘前市の行政境界で水田のデータを切り抜きます。これは
まず水田のデータ(入力ベクタレイヤー)を選びます。つぎにクリップレイヤーとして弘前市の行政データ(ポリゴン)を選びます。すると、弘前市のみの水田データになります。
完成した弘前市のリンゴ畑(ピンク色)と水田(黄緑)マップです。なにも複雑なことはしていませんが、実にたいへんな作業でした。

Rでコルモゴロフ・スミルノフ検定
やや古典的なひびきの統計手法ですが、分布の正規性や、2標本の代表値や分布の違いを検定するときに使います。2標本の場合は代表値(一般には平均値でしょうか)が同じなら分布の検定になるという関係だそうです。詳細はほかのウェブにもっと正確な文章が出ているので、ここではRでの検定手法だけメモしておきます。
1)1試料のとき、ここでは正規性の検定をする場合を書きます。
まず、データの読み込みです。「ksdata」という名前でこれから検定するデータを読み込ませます。Rを起動して直接入力する場合です。
>ksdata<-c(90,126,232.41,94.5,156,120.9,180.4,32.25,29.2,100.11,21,23.1,77,78.75,122.32,59.78,95.59,124.95,112.8,98.4,31.39,31.05,26.4,184,72.8,62.98,23.56,53.35,49.5,33.48,99.33,39.2,11.34,31.92,41.8)
このようにカンマ区切りで指定すればよいです。
続いて検定。
> ks.test(ksdata,"pnorm",mean=mean(ksdata),sd=sd(ksdata))
pnormが正規分布のときに使用するコードです。正規分布は平均と
標準偏差(sd)を指定すれば形が書けるので、それぞれksdataのものを使うのだと指定しています。結果は次の通り。p値が0.567なので、有意水準5%とすれば正規分布といってよいかと思います。
One-sample Kolmogorov-Smirnov test
data: ksdata
D = 0.1284, p-value = 0.567
2)2試料のとき
上記のksdataと比較するデータセットをもうひとつ読み込ませます。ks2dataと
しました。
>ks2data=c(94.4,117,230.4,91,166.32,139.12,206.48,56.76,29.6,96.75,19.2,23.18,104,82.5,115.2,65.52,100.33,113.9,132.8,96,29.4,49.92,26.8,149.73,74.52,61.36,23.4,39.22,49.02,49.02,33,102,33.44,34.04,34.79)
2試料を比較します。
> ks.test(ksdata,ks2data)
結果は次の通り。
Two-sample Kolmogorov-Smirnov test
data: ksdata and ks2data
D = 0.1143, p-value = 0.9763
alternative hypothesis: two-sided
警告メッセージ:
In ks.test(ksdata, ks2data) :
タイがあるため、正確な p 値を計算することができません
タイとは同一値のデータとのことです。なんだか不安を感じるメッセージがあるものの、とりあえずp=0.9763なので2つの分布に差はないとしてよいかと思います。それぞれの平均も下記のとおりです。実はクロテンの左右の精巣サイズのデータでした。片方が人間だと少し下がっているとかいいますが、たぶんサイズはそれほど違わないのではないでしょうか!
> mean(ksdata)
[1] 79.07314
> mean(ks2data)
[1] 82.00343
Rでの一般化線形混合モデル(GLMM)解析、どのモデルを使うか
ところで、GLMMについての説明をとてもおおざっぱにすると、芯になる分布を決めておき、さらに個体差とか場所による差をモデルに組み込んで実際のデータにあてはめるという解析です。芯となる分布の形は次のようなものがあります。
0,1データ:ベルヌーイ分布(二項分布の特殊なケース)
0,1,2,3,…N(上限がある)というデータ:二項分布
0,1,2,3,…(上限がない)というデータ:ポアソン分布
0以上の連続値のデータ:ガンマ分布
連続値のデータ:正規分布
どのような分布にあてはめるのが最もよいか、個体差や場所差を組み込んだほうがよいのか、などを吟味することになります。その際にはAIC(赤池の情報化基準)を用います。
Rでの一般化線形混合モデル(GLMM)解析、はじめの一歩、つづき
RとパッケージglmmMLがそろったらデータを用意します。サンプルデータが北大の久保さんのウェブサイトにあり、岩波書店から出版されているご著書(7.4.1の章)と比べながら作業できます。(下記ウェブサイトからサンプルデータをダウンロード可能)
生態学データ解析 - 本/データ解析のための統計モデリング入門
なお、RではExcelのCSVデータを読むことができます。サンプルデータをわかりやすいところに保存しておいたら、Rを立ち上げてコンソールに次のように打ち込みます。
>d<-read.csv(file.choose())
このdは読み込ませたデータセットにつける名前です。なので、違う名前でもよいわけで、下記ではdata1とつけています。また、コンソールのあとに「<ー」となっていますが、イコールと同じ意味です。慣れている人は上記のfile.chooseというコマンドを使わないでできるようですが、初心者にはこのコマンドが使いやすいのでした。しかし、フォルダやファイル名は英名にしておいたほうが無難だと思います。
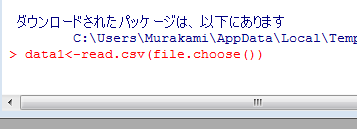
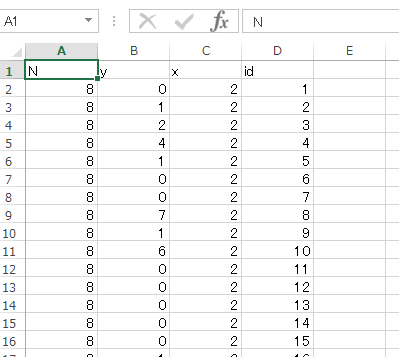
するとどのファイルを読み込ませるか、指定できますので先ほど保存しておいたフォルダに行って、ファイルを選べばOKです。久保さんのサンプルデータは次のようになっています。idの列は個体番号を示しているようですが、このままではただの数値になってしまうので指定する必要があります。このあたりは久保さんの本に丁寧に解説されています。
Rでの一般化線形混合モデル(GLMM)解析、はじめの一歩
生態学の分野では統計解析で一般化線形モデル(GLM)や一般化線形混合モデル(GLMM)が利用されるようになっています。実際に解析をはじめる際に、ちょっとしたことでつまずいてきた経験からやはり備忘録を残しておきます。(コンピュータに詳しい人にとって、とても当たり前の話が私のような初心者にはハードルになるのです!)
Rのダウンロードはさすがにできているという前提で進めます。GLMの分析ツールはRにデフォルトで組み込まれているようですが、GLMMは入っていないので別途専用のパッケージ(glmmML)をダウンロード、インストールする必要があります。これはRを立ち上げてダウンロードするのが確実です。
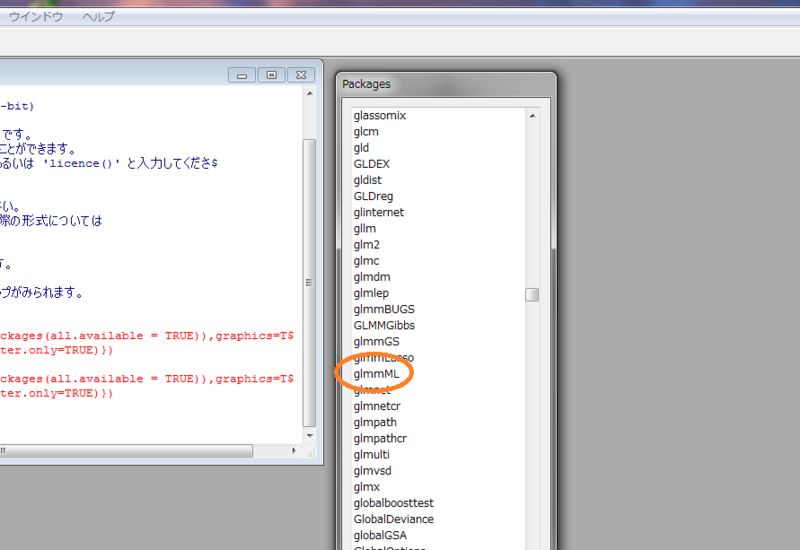
まず、Rのメニューからパッケージタブを選び、「CRANミラーサイトの設定」をクリックします。これはパッケージをダウンロードしてくる相手先の設定です。JAPAN(TSUKUBA)などでよいです。次にあらためてパッケージ>パッケージのインストールを選ぶと下記のようにポップアップの中にglmmMLが登場するのでこれを選べばOKです。
ティルトレンズによる流氷の写真



下の本の中にミニチュア風の写真というのがあり、ちょっと気になっていました。私たちの町では流氷が接岸したので、せっかくなのでこのミニチュア風写真にチャレンジしました。
Amazon.co.jp: 深味のある写真を撮るためのフォトクリエイティブテクニック: デイビッド・ドゥチェミン, パセイジ: 本
この本ではティルトレンズという特殊なレンズを使用していますが、まともに買うと10万近くするような高価なものです。そこでネットを調べてみると引伸し機のレンズで代用できるとのこと。さっそくオークションで引伸し機レンズ(EL-NIKKOR 50mm F4)を購入しました。問題はカメラへの接続です。理想的には蛇腹のようなもので固定できればよいですが、ゴム手袋で代用することもあるそうです。手元にあったのはスキーで寒いときに顔を覆うゴムマスク。ちょうどよいことに鼻の部分が空いていて、レンズを差したらなんとなくはまりました。レンズまわりを輪ゴムでとめて完成。OLYMPUSフォーサーズだと画角が半分なので少し望遠気味になりますが、問題なく使用できました。もちろん露出はマニュアルです。


